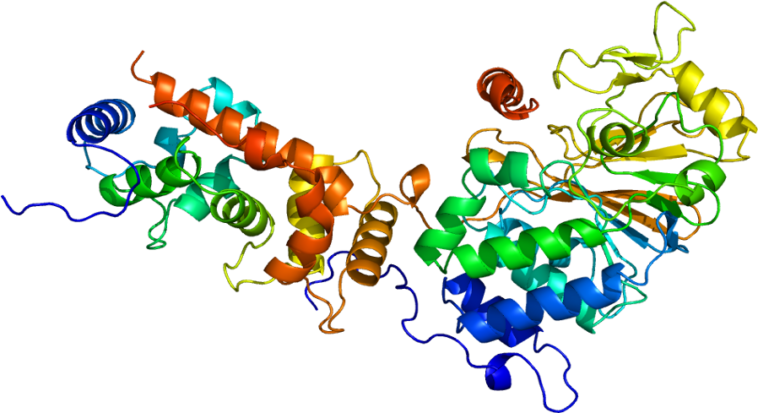
RoseTTAFold, une nouvelle IA inspirée par AlphaFold réussit à prédire la structure des protéines plus complexes.
Les protéines jouent un rôle clé dans l’organisme et principalement en ce qui concerne les cellules. Pour différentes recherches, la compréhension des structures des complexes protéiques est essentielle. Toutefois, prédire ces structures n’est pas une tâche facile ni rapide. Mais désormais, des solutions d’IA facilitent le travail des scientifiques.
De AlphaFold à RoseTTAFold : une IA pour prédire la structure des protéines
Une protéine est composée de longues chaînes de plusieurs acides aminés entrelacés qui s’avèrent très difficiles à déchiffrer. Depuis des dizaines d’années, la prédiction de ces structures est une tâche ardue pour les scientifiques. Cependant, il s’agit d’une étape primordiale pour comprendre le fonctionnement de la vie.
Il n’y a pas très longtemps, des chercheurs ont démontré qu’en utilisant l’intelligence artificielle, il était plus facile de les prédire avec précision. Récemment, une nouvelle approche plus exceptionnelle appelée RoseTTAFold a été développée. À son origine se tient une équipe dirigée par David Baker et Minkyung Baek de l’Université de Washington à Seattle. En effet, toujours basée sur l’IA, leur méthode fonctionne sur des complexes protéiques, en plus des simples protéines.
Les programmes comme RoseTTAFold, ou AlphaFold de DeepMind, réduisent considérablement le temps nécessaire à la prédiction de la structure des protéines. Sans l’intervention de l’IA, cela prend des années aux scientifiques, mais avec ces technologies, il ne faut que quelques jours.
Une meilleure approche ?

Ayant remporté le concours CASP en 2020, les résultats obtenus par AlphaFold2 étaient considérés comme extraordinaires. Baker a alors déclaré que leur travail a été inspiré par les avancées de DeepMind. Autrement dit, RoseTTAFold peut également créer des modèles à partir d’une grande base de données, en s’améliorant au fur et à mesure de son apprentissage.
Néanmoins, la différence entre AlphaFold et RoseTTAFold concerne le nombre de pistes explorées par les programmes pour prédire la structure des protéines. Si l’IA de DeepMind n’en comporte que deux, celle de Baek et Baker en utilise plusieurs. En termes de précision, AlphaFold2 reste le meilleur, mais RoseTTAFold permet de prédire la structure des complexes multiprotéiques.
Quoi qu’il en soit, ces avancées technologiques contribuent à améliorer la compréhension de la biologie structurelle. Selon DeepMind, c’est en effet l’objectif commun de toutes ces recherches. De plus, le fait que Baek et Baker ont publié leur code informatique sur le web le confirme sans doute.
- Partager l'article :